
2023年3月7日,来自俄罗斯科学院托木斯克国家医学研究中心癌症研究所癌症进展生物学实验室、托木斯克国立大学转化细胞与分子生物医学实验室、俄罗斯人民友谊大学分子与细胞医学研究所单细胞生物学实验室等机构的科学家们联合在线发表了题为“Comparison of the Illumina NextSeq 2000 and GeneMind Genolab M sequencing platforms for spatial transcriptomics”的研究成果。该研究基于40001百老汇官网电子游戏的GenoLab M和Illumina的NextSeq 2000两个测序平台分别对3例卵巢癌样本进行10x Genomics Visium空间转录组测序,并对得到的测序数据进行比较分析。研究发现GenoLab M在测序数据质量方面的表现与NextSeq 2000不相伯仲。在后续的降维分析、细胞亚群分析及差异表达基因方面也与NextSeq 2000分析结果均保持了高度的一致性,证明其应用于空间转录组的实力。
背景介绍
单细胞转录组测序(scRNA-seq)是在单个细胞水平进行高通量转录组测序的一项新技术,能够有效解决细胞异质性以及组织转录组测序(bulk RNA-seq)被掩盖的细胞群内转录组异质性难题。该技术有助于发现新的稀有细胞类型,深入了解细胞生长与分化过程中的表达调控机制。然而,在进行scRNA-seq测序的细胞解离过程中不仅存在与细胞应激、细胞死亡/细胞聚集等相关的阻碍因素,同时还会丢失空间信息。此外,一些组织中的特定细胞类型,尤其是免疫细胞,难以从组织中解离出来,更限制了scRNA-seq测序的广泛应用。技术更迭,空间转录组技术横空出世,更是在2020年被Nature Method评为年度技术。其中基于10X 单细胞平台的空间转录组技术成为了主流。该技术不仅能够检测完整组织切片的总mRNA,而且还能将组织的空间信息和形态学内容与mRNA整合在一起,最终绘制出基因表达的空间位置信息。为研究细胞功能、细胞表型和组织微环境等提供了关键的信息。
40001百老汇官网电子游戏的GenoLab M测序仪作为一个新的测序平台,在转录组与lncRNA测序、WGS和WES方面表现优异,是否可以应用到空间转录组呢?对此,研究使用3份卵巢癌样本,分别在GenoLab M与NextSeq 2000上完成空间转录组测序,比较两个平台测序结果的一致性。
结果概要
01实验设计与测序质量
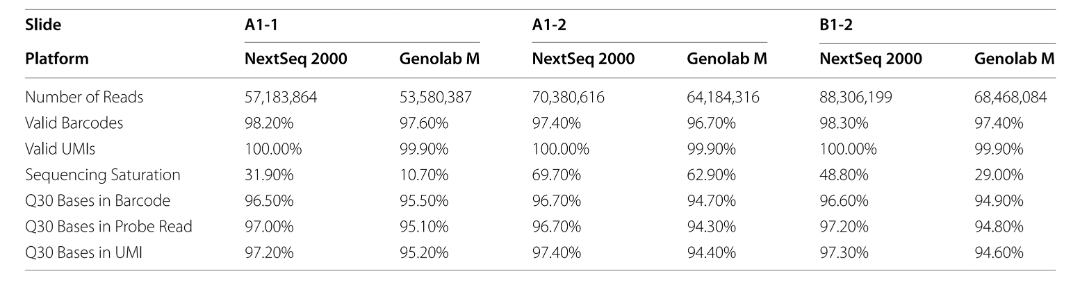 表1 三例卵巢癌样本两个测序平台的测序质量比较
表1 三例卵巢癌样本两个测序平台的测序质量比较
本研究将3份卵巢癌的石蜡切片样本,分别构建10xGenomics Visium的测序文库。随后将3个文库一分为二,分别使用GenoLab M与NextSeq 2000测序平台进行测序。2个平台同一个样本测序数据总量接近,NextSeq 2000中有效barcode的百分比和有效UMI的百分比分别高出GenoLab M 0.74%和0.1%。而Q30碱基的百分比在barcode、探针和UMI对比中也比GenoLab M分别高出1.6%、2.2%和2.6%。以上的差异主要是GenoLab M测序数据深度与测序reads duplication相对较低。
* Barcode是含有空间位置标签的一段核酸序列,区分不同的spots,每个spots包含数十个细胞用于定位;
* Probe探针是用于结合mRNA的的序列;
* UMI是区分不同mRNA,用于绝对定量的核酸序列;
02reads mapping与基因表达绝对定量
两个平台组织的spots下reads的比例基本一致,差异不显著。每个spots和组织的平均reads数与测序深度也基本一致。除样本B1-2外,GenoLab M平台中每个spot的UMI计数和基因的中位数以及检测到的基因数都比NextSeq 2000高。而NextSeq 2000在Reads Mapped Confidently to Probe Set 的表现中略高于GenoLab M。这与探针reads更高的测序质量一致。尽管如此,在Reads Mapped Confidently to the Filtered Probe Set的对比中 GenoLab M仍然有比NextSeq 2000更好的表现。
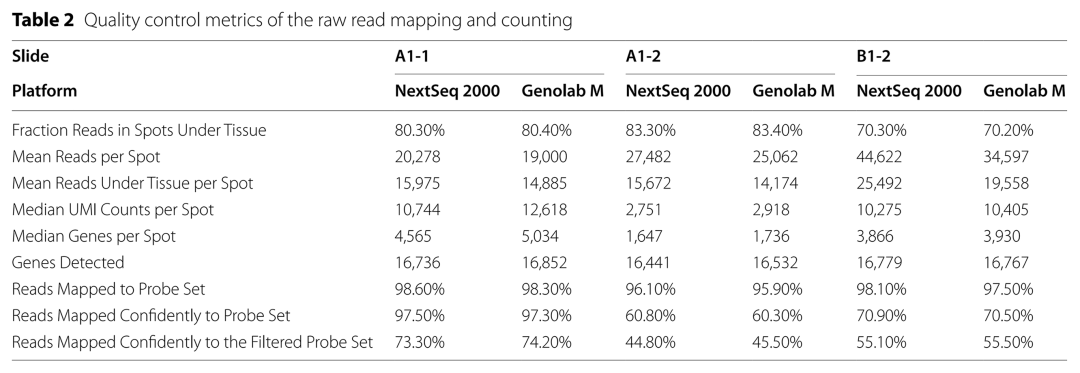 表2 reads mapping和UMI计数的测序质量矩阵比较
表2 reads mapping和UMI计数的测序质量矩阵比较
03基因、UMIs、reads在组织的spots上的检测情况
两个测序平台组织的spots下总的UMI和基因数目具有很高的一致性(Figure 1A)。三个样本的基因-UMI关系在两个平台的一致性也很高(Figure 1B)。此外,GC谱也高度一致(Figure 1C),说明两个测序平台都没有明显的测序偏向性。
接着研究者去除低表达基因和基因数目较少的spots后,比较两个平台的基因交并集情况。结果表明平台特有基因相对于交集占比都比较小,其中GenoLab M的特有基因稍多(Figure 1D)。在两个平台上测序的spots基因交集之间存在很强的相关性。三个样本A1-1、A1-2 和 B1-2 样本的平均相关系数分别为 0.82、0.90 和 0.99(Figure 1E)。
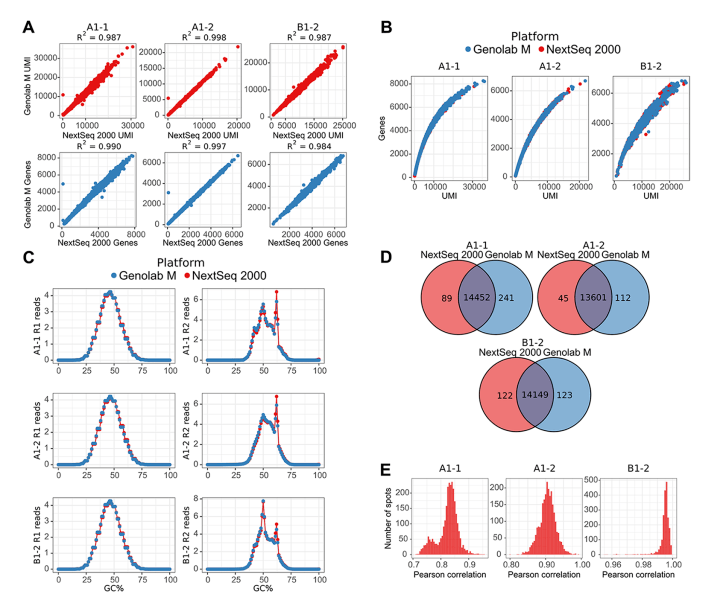 图1 两个测序平台检测到的reads数目,UMI和基因的比较
图1 两个测序平台检测到的reads数目,UMI和基因的比较
A 组织spots上转录本(基于UMI计数)和基因数的一致性分析;
B 组织spots上基因的UMI分布;
C reads的GC含量谱;
D 平台间共有和特有基因的维恩图;
E SCT转化基因交集的Pearson相关系数分布;
04降维、细胞亚群聚类和差异表达分析
三个样本在两个平台的测序结果分析后得到的细胞亚群几乎一致,都是16个亚群(Figure 2A),来自不同测序平台分析的样本的组织spots所在的位置也非常接近(Figure 2B)。A1-1含有7个亚群、A1-2有4个亚群、B1-2有5个亚群。差异表达基因(DEG)检测方面,NextSeq 2000和GenoLab M分别检测到的特有DEG约占所有检测的DEG的16%;共有的占比更高为68%(Figure 2C-E)。
* DEG的筛选条件,FDR<0.01且log fc=''>0.25(差异倍数>1.19);
* FDR False Discovery Rate错误发现率;
* LTF Log2(Fold Change) 差异倍数fold change的log2值;
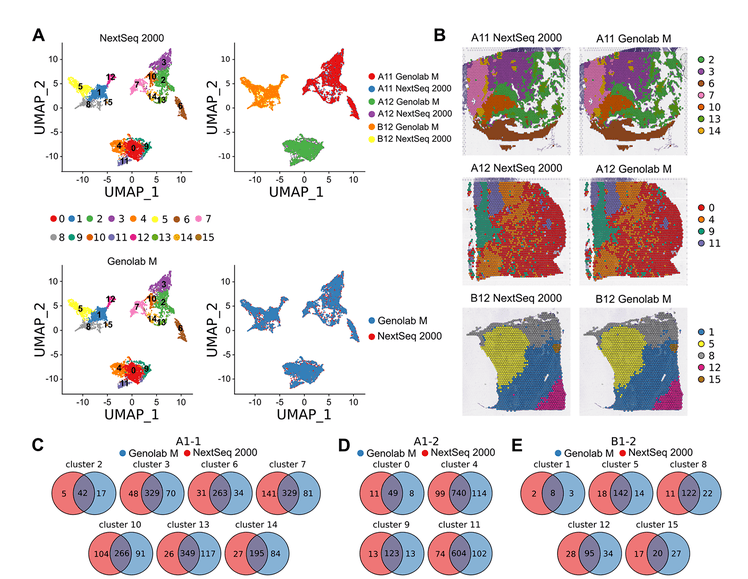 图2 非线性降维、聚类和差异表达分析
图2 非线性降维、聚类和差异表达分析
A 未做批次校正的两个平台的测序数据UMAP降维和聚类;
B 亚群的空间分布;
C A1-1样本的平台间共有与特有差异表达基因的维恩图;
D A1-2样本的平台间共有与特有差异表达基因的维恩图;
E B1-2样本的平台间共有与特有差异表达基因的维恩图;
05差异表达基因的深入分析
研究对平台共有与特有的DEG深入分析以评估特有DEG的来源。通过DEG的计数来可视化LFC和FDR的分布。图3A展示了DEG-LFC在组织spots亚群聚类的分布,发现所有平台特有的DEG的LFC均分布在所选阈值的边缘。图3B展示了DEG-FDR在组织spots亚群聚类的分布,平台特有DEG的FDR值往往最大(代表显著性值最低)。综合来说,平台特有DEG比较接近阈值,而共有的DEG则具有较小的FDR和较高的差异倍数。
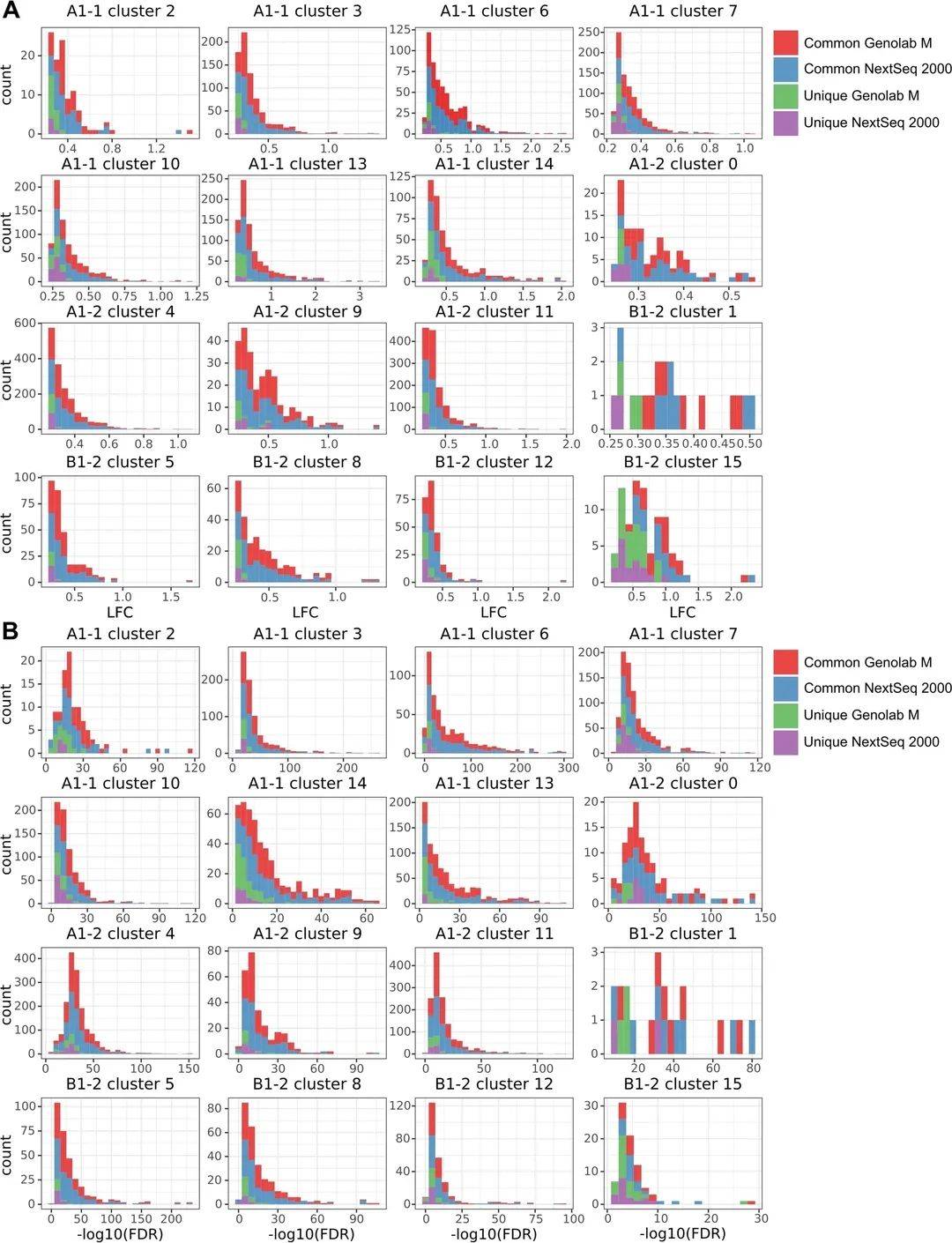 图3 亚群差异表达的差异倍数(A)与FDR(B)的分布情况
图3 亚群差异表达的差异倍数(A)与FDR(B)的分布情况
结论
研究首次比较了GenoLab M和NextSeq 2000在空间转录组测序的性能,两个平台均采用了可逆终止的边合成边测序的技术。三份Visium的测序文库在两个测序平台上得到了一致的空间转录组分析数据。研究结果表明GenoLab M的测序性能与NextSeq 2000接近,适用于进行基于10 × Genomics的空间转录组测序。